変異の絞り込み 【1】論文紹介
変異の絞り込み 【2】変異検出
変異の絞り込み 【3】候補の絞り込み方
変異の絞り込み 【4】公開データベースを用いた候補の絞り込み
変異の絞り込み 【4.5】お詫びと訂正
変異の絞り込み 【5】変異のクオリティとインパクト
変異の絞り込み 【6】遺伝型による絞り込み
前回までは父、母、子(IV-1)のトリオのexome解析により、疾患関連変異候補を24個にまで絞り込みました。
今回は、子の同胞(兄弟)のexomeシーケンスデータを用いて、より絞り込みます。

まず、同胞(IV-2、IV-3)のexomeシーケンスデータから変異を検出します。
次に、前回までで絞り込んだ24個の変異が存在する24遺伝子上にある3人の子どもの変異を抜き出します。
さらに、再びQmergeVCFを使い、3人の変異情報を統合します。トリオの解析ではLCA罹患者である子(IV-1)をcase、非罹患者である両親をcontrolと設定しましたが、今回は罹患者IV-1、IV-3をcase、非罹患者であるIV-2をcontrolと設定しました。
その結果、疾患関連候補の24遺伝子上に、case2人に共通する変異133個が検出されました。
このあとは、トリオとほぼ同じように、公開データや変異のクオリティによって絞り込みました。
最後の、遺伝型による絞り込みにおいては、LCA罹患者であるIV-1、IV-3はホモ、非罹患者のIV-2はヘテロまたは野生型の変異を抜き出しました。

なんと、疾患関連変異候補はたったの4個にまで絞り込まれました。
問題は、この4個にLCAを引き起こしている変異が含まれているかどうかです。
残った4つの疾患関連変異候補はこちらです。
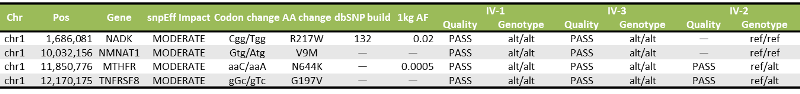
4個の中に、NMNAT1の9番目のバリンをメチオニンに置換する変異(上から2つめ)が含まれており、元々このexomeシーケンシングデータを公開したFalkらの報告と一致しました。
この結果は前回の記事の総括を非常に強く支持するものになりました。
以上、全7回にわたり、Exomeシーケンスデータから検出した変異から、重大な疾患に関する変異の候補をどのように絞り込むかについて連載いたしました。
(一部自社製ソフトを使用していますが、方針としては汎用性があるものです)
本解析ではトリオだけでなく罹患同胞の情報も用いたことで、非常に精度の高い絞り込みが行えました。
他にも、今回用いているdbSNPや1000人ゲノムプロジェクトなどの公開データがより充実することや、人種や民族別のデータベースがあると、精度が上がると考えられます。
希少な遺伝性の疾患の原因探索や治療には、変異を検出した後のフィルタリングが重要ですが、今後データベースなどの発展や充実によって、遺伝性疾患の研究が進展していくといいですね。