今や泣く子も黙るシングルセルRNA-seqの兄弟分、シングルセルATAC-seqをご存じでしょうか?
その名の通り、クロマチンアクセシビリティを調べるATAC-seqを、1細胞レベルで行う手法です。
bulk ATAC-seqと違って高分解能で見ることができ、シングルセルRNA-seqと違ってエピジェネティックな制御を見ることができます。
がん組織など様々な細胞が混在したサンプルを扱う分野で盛り上がってきている手法で、
シングルセルRNA-seqと組み合わせて使われるケースがよく見られます。
そんなシングルセルATAC-seqは、現在どんなDRY解析が行われているのでしょうか?
論文を調べたり、最前線の研究者の方々のお話を聞いてしばしば見られたフローは、
10X Genomics Chromium Systemで得たシーケンスデータに対し
Cell Ranger ATACでピーク検出まで行い、Loupe Cell Browserで可視化する、というものでした。
シングルセルRNA-seq同様のクラスタリング結果のplotと、クラスタごとのピーク位置がわかります。
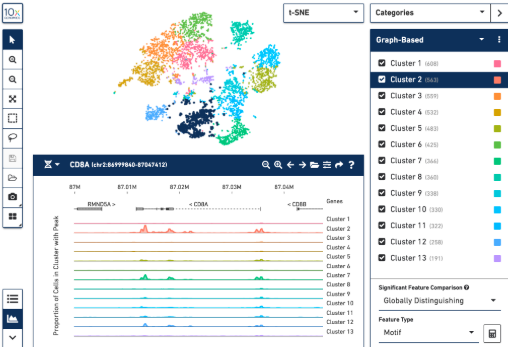
※10X Genomics ホームページより
Loupe Cell Browserでは結果をRNAの結果と見比べることもできます。
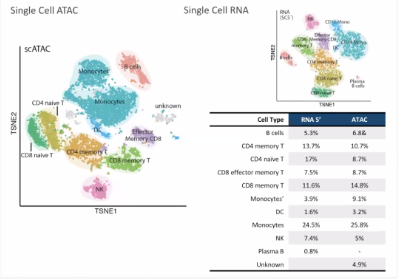
※10X Genomics主催ウェビナー資料より
ただし、RNAとATACを全く同じライブラリについて行えるわけではない(アダプタの構成が異なるため)ので、そこは注意ですね。
bulk ATAC-seqだと、ここから高次解析としてde novo モチーフ検索を行うなどします。
しかしシングルセルの方は少なくとも現在、高次解析よりも、他の解析とのデータ統合結果をどう見るか、ということが注目の的のようです。
以上、最新シングルセルATAC-seqレポートでした。
ちなみに、昨今のシングルセル業界ではATACの他に、細胞表面にどんなタンパク質が発現しているのか調べるCITE-seq、
Nanoporeのロングリードシーケンス結果を使用するRAGE-seqなどを、研究目的に応じて組み合わせることがあります。
それらの手法についてはまたいずれ。