こんにちは、iijm_lです🐰
Pythonでバイオインフォマティクス、といえば、Biopythonですね。
Biopythonには便利な機能が多く存在します。
SeqIO や SeqRecord のような配列データ操作系はよく使用するのですが、
他のモジュールについては、まだまだ知らないものが多いです。
便利なツールを使いこなして、できることの幅を広げたいということで、
🐰「ブログを通して、Biopythonとお友達になろうっ!!!!」
と、やや大きめの個人目標を掲げておきます。
今回は、Biopythonで分子系統樹の作成 を目標にしました✨
やりたいこと
以前「ヒトとサルは遺伝子的にそんなに近くない」という話を思い出し、
ヒトに最も近い霊長目選手権を開催することにしました。
具体的なフローは、以下の通りです。
- 霊長目の multi FASTAファイルを用いて、アライメントを行う。
- アライメント結果を用いて、分子系統樹を作成する。
ここで、バイオ用語のおさらいをしておきましょう。
- アライメント
DNAやRNA、タンパク質などの配列同士を比較し、類似性を算出します。
これにより、配列モチーフに基づく機能予測や、生物種群の特定、進化系統の推測など、多くの情報を得ることができます。 - 分子系統樹
生物の配列情報の類似性を表した図です。
枝分かれした先に、その生物種やタンパク質名が記載されます。
枝の長さが形質の差分を表しており、類似性が高いほど近くに、低いほど離れて描かれます。
使用データ
NCBI Datasets ページより、霊長目の Cytochrome c oxidase subunit I (COX1) の塩基配列を取得しました。
→ Datasets - NCBI
カレントディレクトリにprimates_COX1.fnaとして配置しました。
$ cat primates_COX1.fna >Homo_sapiens ATGTTCGCCGACCGTTGA... >Macaca_fuscata ATGCTCATCAATCGCTGA... ...
参加選手(生物種)12名の紹介です。
- ヒト (Homo sapiens)
- ニホンザル (Macaca fuscata)
- アカゲザル (Macaca mulatta)
- チンパンジー (Pan troglodytes)
- ゴリラ (Gorilla gorilla gorilla)
- ボルネオオランウータン (Pongo pygmaeus)
- マントヒヒ (Papio hamadryas)
- マンドリル (Mandrillus sphinx)
- コモンマーモセット (Callithrix jacchus)
- コモンリスザル (Saimiri sciureus)
- スローロリス (Nycticebus coucang)
- ワオキツネザル (Lemur catta)
※ 昨今は多様性の時代のため、ヒトに近くなさそうな生物種も参加しています。
環境
- CentOS 7
- Python 3.7.10 (Anaconda)
- Biopython 1.78
- matplotlib 3.3.4
アライメント
配列アライメントデータを操作できるBio.Alignを使います。
Bio.Align.Applicationsモジュールを使用すると、
ローカル環境にある配列アライメントツールを呼び出し、pythonから簡単に実行できます。
今回は、一般的なアライメントツールであるClustalWを使います。
他にも、MafftやMuscleなどが使えます。
※ 事前に以下のURLより、ClustalW2をダウンロードし、パスを通してください。
http://www.clustal.org/download/current/
>>> from Bio.Align.Applications import ClustalwCommandline >>> cmd = ClustalwCommandline("clustalw2", infile="primates_COX1.fna") >>> print(cmd) clustalw2 -infile=primates_COX1.fna >>> stdout, stderr = cmd()
これで、ClustalWの実行が完了です。 アライメント結果として、2つのファイルが作成されました。
$ ls . primates_COX1.fna # 入力ファイル primates_COX1.aln primates_COX1.dnd
分子系統樹の作成
アライメント結果の距離行列やノード情報を操作したり、
分子系統樹の描画ができるBio.phyloを使います。
>>> from Bio import Phylo >>> tree = Phylo.read("primates_COX1.dnd", "newick") >>> Phylo.draw(tree)
分子系統樹が表示されました。
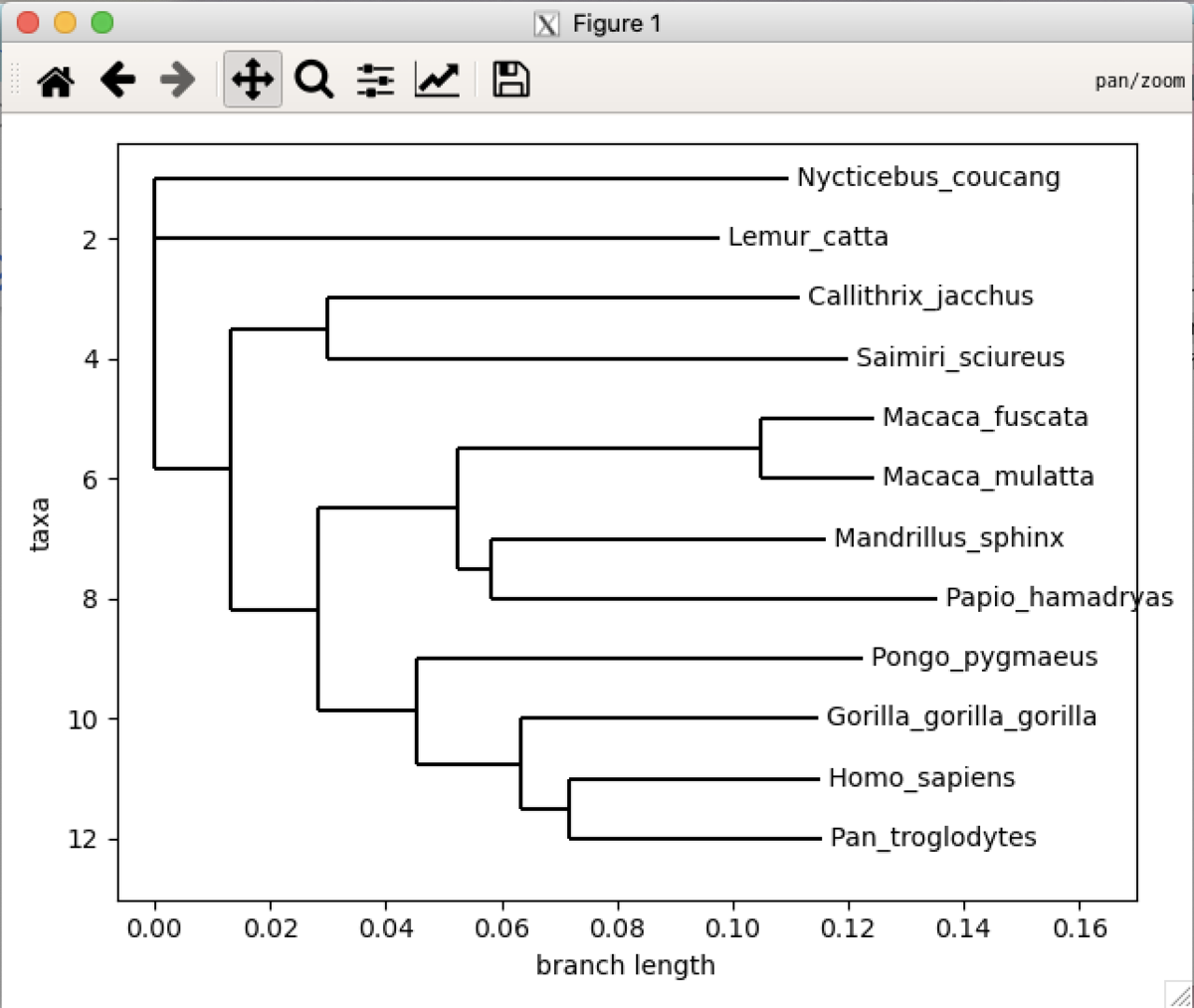
COX1の塩基配列において、ヒト(Homo sapiens)に最も近しいのは、
チンパンジー(Pan troglodytes)であることが分かりました。
一般的にサル(monkey)と呼ばれるニホンザル(Macaca fuscata)やアカゲザル(Macaca mulatta)は、ヒトからかなり離れていますね。
それよりも、ゴリラ(Gorilla gorilla gorilla)やボルネオオランウータン(Pongo pygmaeus)の方がヒトに近いんですね🤔
系統樹の描画に話を戻します。
図ではなく、ASCIIを使ってコンソール上に表示することも可能です。
>>>tree = Phylo.read("primates_COX1.dnd", "newick") >>>Phylo.draw_ascii(tree) _________________________________________ Nycticebus_coucang | |____________________________________ Lemur_catta | _| ______________________________ Callithrix_jacchus | _____| | | |__________________________________ Saimiri_sciureus | | | | ______ Macaca_fuscata |____| ___________________| | | |______ Macaca_mulatta | _________| | | | _____________________ Mandrillus_sphinx | | |_| |____| |_____________________________ Papio_hamadryas | | _____________________________ Pongo_pygmaeus |______| | ___________________ Gorilla_gorilla_gorilla |______| | ________________ Homo_sapiens |__| |________________ Pan_troglodytes
まとめ
今回は、pythonでアライメントと分子系統樹の描画を学びました😃
BioPythonとお友達になれる日は果たして来るのか...?
乞うご期待ください💡