こんにちは、受託コンサルティングチームの hosor です。
先日、気温が 3 ℃ かつ雨の中テニスをしてきました。
さて今回は、染色体上の着目箇所を可視化する R パッケージ chromoMap について紹介していこうと思います。
作業環境
- Windows11
- Rstudio 2023.03.0 Build 386
- R v4.2.3
- chromoMap v4.1.1
使用データ
まず、ヒトの染色体の情報を chr_file.txt に記載しました。
- 1 列目: 染色体名
- 2 列目: 染色体の開始位置
- 3 列目: 染色体の終了位置
染色体の長さは Genome assembly GRCh38 に基づいています。
1 1 248956422 2 1 242193529 3 1 198295559 4 1 190214555 5 1 181538259 6 1 170805979 7 1 159345973 8 1 145138636 9 1 138394717 10 1 133797422 11 1 135086622 12 1 133275309 13 1 114364328 14 1 107043718 15 1 101991189 16 1 90338345 17 1 83257441 18 1 80373285 19 1 58617616 20 1 64444167 21 1 46709983 22 1 50818468 X 1 156040895 Y 1 57227415
次に、染色体上の着目箇所の情報を anno_file.txt に記載しました。
今回は dbSNP にて risk factor[Clinical Significance] と検索した結果から、適当に 10 SNP 選びました。
- 1 列目: 要素 (ID, シンボルなど)
- 2 列目: 染色体名
- 3 列目: 染色体の開始位置
- 4 列目: 染色体の終了位置
rs268 8 19956018 19956018 rs662 7 95308134 95308134 rs671 2 58723690 58723690 rs699 1 230710048 230710048 rs4880 6 159692840 159692840 rs4961 4 2904980 2904980 rs4994 8 37966280 37966280 rs5443 12 6845711 6845711 rs5491 19 10274864 10274864 rs5848 17 44352876 44352876
スクリプト
chr_file = "chr_file.txt" anno_file = "anno_file.txt" chromoMap(chr_file, anno_file)
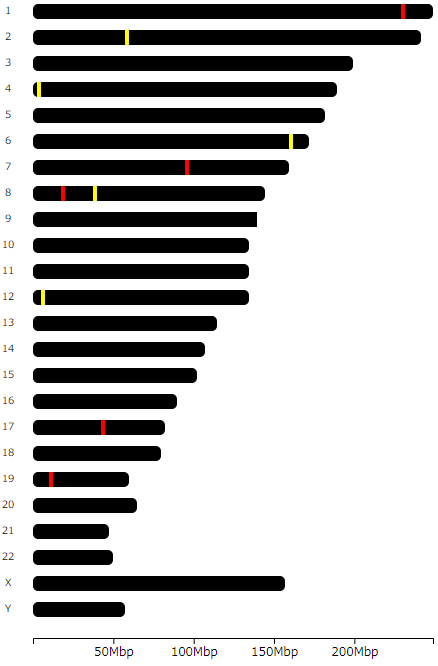
出力結果は以下のようになります。

各 SNP が染色体上のどこに位置しているのか、一目瞭然ですね!
例えば、5 列目に何らかのカテゴリ情報を加えた anno_file_category.txt を作成すると、カテゴリごとに色分けした図を描画することもできます。
※ A, B のカテゴリに特に意味はありません。
rs268 8 19956018 19956018 A rs662 7 95308134 95308134 A rs671 2 58723690 58723690 B rs699 1 230710048 230710048 A rs4880 6 159692840 159692840 B rs4961 4 2904980 2904980 B rs4994 8 37966280 37966280 B rs5443 12 6845711 6845711 B rs5491 19 10274864 10274864 A rs5848 17 44352876 44352876 A
anno_file_category = "anno_file_category.txt" chromoMap(chr_file, anno_file_category, data_based_color_map = T, data_type = "categorical", data_colors = list(c("red", "yellow")))
chromoMap のドキュメントでは、他の機能についてもかなり細かく説明されているので是非ご参照ください。
