こんにちは、システム開発チームの adachit です。
いよいよ連載の最終回となりました。
第4回テーマは「実践:Nextflowのパイプラインをカスタマイズする」です。
Nextflowの長所「自分でパイプラインをカスタマイズする」実例を挙げながら解説します。
カスタマイズの流れ
Nextflowの大きな長所の一つは、自分でパイプラインをカスタマイズできる点にあります。
そこで今回は「nf-core」の「rnaseq」に標準では含まれていない「ヒートマップを描く機能」を追加するカスタマイズの例をご紹介いたします。
オリジナルの「rnaseq」は5個のコンテナから構成されているため、ここに6個目のコンテナを追加する形で、カスタマイズしてみたいと思います。
まず、カスタマイズの大まかな手順は以下の通りです。
①ソースコードのダウンロード
②解析プロトコル(Rスクリプト)の作成
③解析環境(Dockerイメージ)の作成
④ヒートマップ描画用Nextflowスクリプトの作成
⑤「nf-core」のNextflowスクリプトの編集
①ソースコードのダウンロード
「rnaseq」のソースコードをダウンロードするためには、nextflow clone nf-core/rnaseqコマンドを実行します。
実行後、現在のディレクトリに「rnaseq」ディレクトリが作成され、その中にソースコードが格納されます。
②解析プロトコル(Rスクリプト)の作成
ヒートマップを描くための解析プロトコルをRで作成します。
以下はそのスクリプトの一例です。
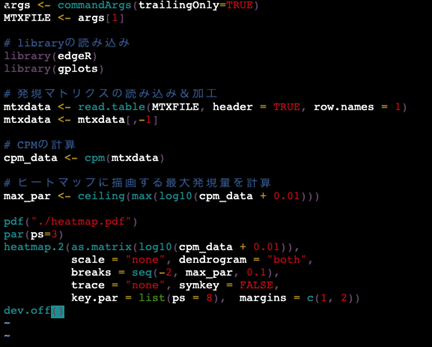
上記は、gplots パッケージに含まれるheatmap.2関数を使用してヒートマップを描き、dev.off()でPDFとして保存するスクリプトになっています。
③解析環境(Dockerイメージ)の作成
Dockerイメージを作成するため、まずDockerfileを作成します。
Dockerfileとは、Dockerイメージを作成するための設計書のようなものです。
Dockerfileでは、Rの実行環境をベースとして指定し、次にヒートマップの描画に必要なRのライブラリ「BiocManager」「edgeR」「gplots」をインストールします。
以下はそのDockerfileの一例です。
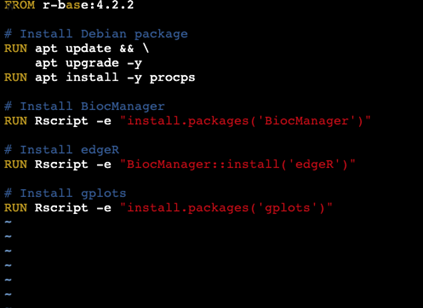
上記のDockerfileを使用して、docker buildコマンドでDockerイメージを作成します。
④ヒートマップ描画用Nextflowスクリプトの作成
ヒートマップを描画するためのNextflowスクリプト「heatmap.nf」を作成します。
以下はそのNextflowスクリプトの一例です。
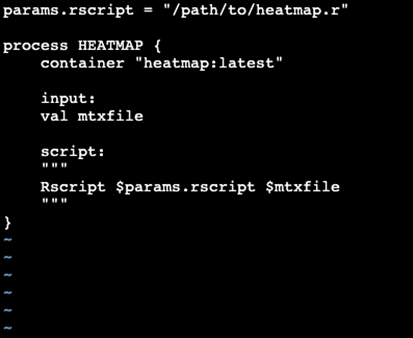
⑤nf-coreのNextflowスクリプトの編集
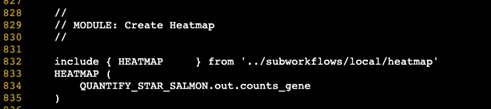
①でダウンロードしたソースコードの中のNextflowスクリプト「rnaseq.nf」を編集します。
このファイルには、オリジナルの1~5番目のブロックを実行するスクリプトが書かれています。
ここに6番目のブロックとしてヒートマップを描画する部分を追加します。
これでカスタマイズは完了です。
nextflow run main.nfコマンドを実行すると、実行ログにヒートマップの描画が記録されていることが確認できます。
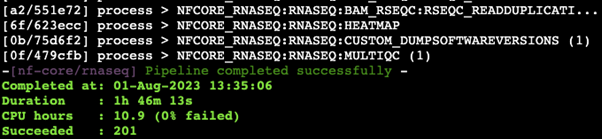
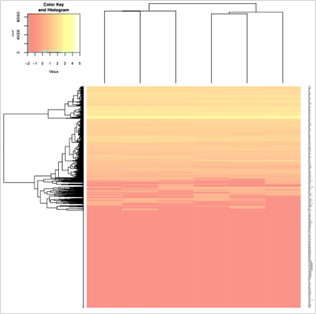
実行結果として、「heatmap.pdf」が正しく出力されました。
以上で全4回連載「Nextflowで始めるNGSデータ解析ワークフロー」は終了となります。
各回の記事は以下の連載リンクから閲覧できます。
※本記事は、2023年8月2日開催の第88回バイオインフォマティクス勉強会「Nextflowで始めるNGSデータ解析ワークフロー」講演内容をベースに作成しております。
動画で本記事の内容を視聴したい、講演資料PDFをダウンロードしたい方は、アメリエフの運営する会員制動画サイト「バイオインフォマティクス実践ラボ」にご登録ください。
