こんにちは、受託コンサルティングチームの hosor です。
シングルセル解析においてクラスタリングを行っている際に、どの resolution が妥当なのか判断できない・クラスタリング結果を包括的に把握したいというケースも多いのではないでしょうか。
そこで今回は R パッケージ clustree を用いて、クラスタリング樹形図 (clustering tree) を描画する方法をご紹介していきます。
クラスタリング樹形図を見ることで細胞がどのクラスタに分類されていくのかが分かるため、データの特徴を理解する際に役立てられると思います。

作業環境
- Ubuntu 22.04.4 LTS
- R v4.3.3
- Seurat v5.1.0
- SeuratData v0.2.2.9001
- clustree v0.5.1
使用データ
ヒト末梢血単核球 (PBMC) 2,700 個を使用しました。
Seurat 公式ページのチュートリアルに従い前処理を進め、Seurat オブジェクト pbmc を作成しました。
スクリプト
まず、例として resolution=0, 0.1, 0.2, ..., 1.0 の 11 パターンでクラスタリングを行います。
参考までに、クラスタリング結果を UMAP 上に反映した図も一部載せます。
for(res in seq(0, 1.0, by = 0.1)){
pbmc <- FindClusters(pbmc, resolution = res)
p <- DimPlot(pbmc, group.by = "seurat_clusters", reduction = "umap") + labs(title = paste0("UMAP_resolution", res))
ggsave(paste0("DimPlot_UMAP_resolution", res, ".pdf"), p, width = 7.5)
}
pbmc を見ると、クラスタリングの結果が RNA_snn_res.XX 列に加えられていることが分かります。
例えば、resolution=0.1 の結果は RNA_snn_res.0.1 列に対応しています。
これを見ることで、各細胞がどのクラスタに割り当てられたのか確認することができます。
head(pbmc, n = 5)
orig.ident nCount_RNA nFeature_RNA seurat_annotations percent.mt
AAACATACAACCAC pbmc3k 2419 779 Memory CD4 T 3.0177759
AAACATTGAGCTAC pbmc3k 4903 1352 B 3.7935958
AAACATTGATCAGC pbmc3k 3147 1129 Memory CD4 T 0.8897363
AAACCGTGCTTCCG pbmc3k 2639 960 CD14+ Mono 1.7430845
AAACCGTGTATGCG pbmc3k 980 521 NK 1.2244898
RNA_snn_res.0 seurat_clusters RNA_snn_res.0.1 RNA_snn_res.0.2
AAACATACAACCAC 0 6 0 0
AAACATTGAGCTAC 0 2 3 3
AAACATTGATCAGC 0 1 0 0
AAACCGTGCTTCCG 0 4 1 1
AAACCGTGTATGCG 0 8 2 2
RNA_snn_res.0.3 RNA_snn_res.0.4 RNA_snn_res.0.5 RNA_snn_res.0.6
AAACATACAACCAC 0 2 2 1
AAACATTGAGCTAC 3 3 3 3
AAACATTGATCAGC 0 2 2 1
AAACCGTGCTTCCG 1 1 1 2
AAACCGTGTATGCG 2 6 6 6
RNA_snn_res.0.7 RNA_snn_res.0.8 RNA_snn_res.0.9 RNA_snn_res.1
AAACATACAACCAC 1 6 1 6
AAACATTGAGCTAC 3 2 2 2
AAACATTGATCAGC 1 1 1 1
AAACCGTGCTTCCG 2 4 4 4
AAACCGTGTATGCG 6 8 7 8
上記の RNA_snn_res.XX 列を参照しつつ、clustree 関数を実行します。
p <- clustree(pbmc, prefix = "RNA_snn_res.")
ggsave("Clustree.pdf", p, height = 10)
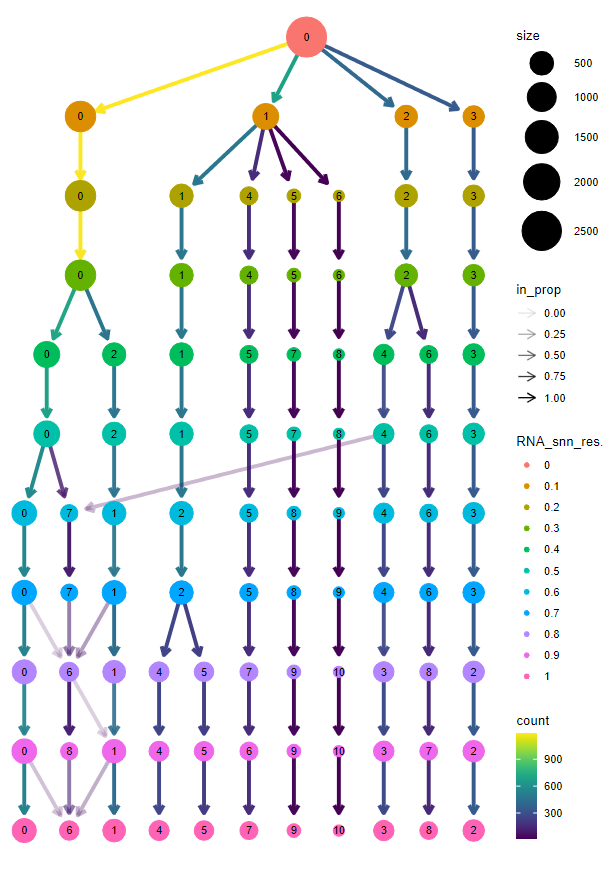
クラスタリング樹形図を描画することができました!
上から resolution=0, 0.1, 0.2, ..., 1.0 の結果に対応しており、丸の大きさや矢印の色・透明度が細胞数を表します。
図より、例えば、以下のことが分かります。
- resolution=0.1 でクラスタ 3 となった細胞群は、resolution=1.0 まで分離しない。つまり、1 つの細胞種であることが考えられる。
- resolution=0.4 の場合はクラスタ数は 9 個、resolution=1.0 の場合はクラスタ数は 11 個であり、resolution の値を大きくしてもあまり変わらない。また、FindClusters 関数の resolution のデフォルト値 (推奨値) は 0.8 *1 であり、resolution=0.8 の場合はクラスタ数は 11 個である。これらのことより、今回のデータに含まれる細胞種は 10 個程度だと推測される。
上記はあくまで一例ですが、さらに広い範囲・細かい範囲の resolution について描画することで、より詳細な結果を得ることも可能です。
シングルセルデータの特徴の理解に役立つと思うので、ぜひご活用ください!
補足
以下の Vignettes を参考にしました。
https://cran.r-project.org/web/packages/clustree/vignettes/clustree.html#seurat-objects
