heatmap関数のRowSideColors/ColSideColorsオプションの紹介です。
gplotsパッケージのheatmap.2やheatmap3パッケージでも同様の機能があります。
ヒートマップのデンドログラムの横に、カラフルなサイドバーを付ける機能です。
テスト用のマトリクスを作ります。
(結果は実行するたびにランダムに変わります)
mat<-matrix(sample(100,70,replace=T),ncol=7)
rownames(mat)<-c("geneA","geneB","geneC","geneD","geneE","geneF","geneG","geneH","geneI","geneJ")
colnames(mat)<-c("sample1","sample2","sample3","sample4","sample5","sample6","sample7")
このマトリクスでヒートマップを作成するとこうなります。
heatmap(mat)

以下は左にあるデンドログラムの横に、サンプルごとに色を付ける場合の例です。
RowSideColorsオプションに、色名(16進数での指定も可)のベクトルを指定します。
この色名のベクトルは、マトリクスの行数と同じ長さである必要があります。
nrow(mat)
[1] 10
また、色はマトリクスの各行に対して指定していますので、ヒートマップではクラスタリングされた順に並びかえられて描画されます。
rownames(mat)
[1] "geneA" "geneB" "geneC" "geneD" "geneE" "geneF" "geneG" "geneH" "geneI" "geneJ" ←この順に色が指定されます
rcol<-c("red","blue","green","goldenrod","violetred","salmon","orange","springgreen","pink","black")[
heatmap(mat,RowSideColors=rcol)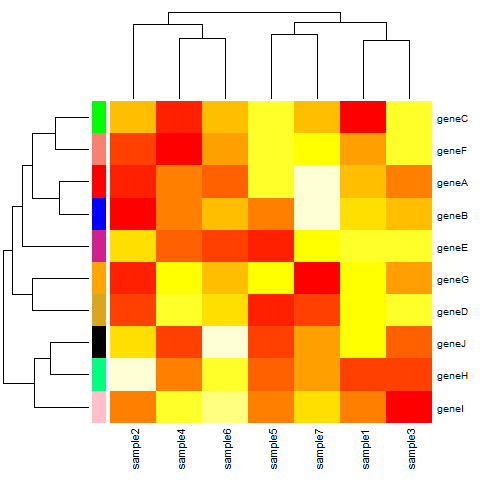
クラスタリングされたグループごとに色をつけたい場合は、マトリクスをあらかじめクラスタリングしたうえでcutree関数を使います。
mat.d<-dist(mat)
mat.h<-hclust(mat.d)
mat.c<-cutree(mat.h,k=3)cutree関数のkで指定した数値と同じ長さの色名のベクトルをRowSideColorsに指定します。
clust.col<-c("red","blue","orange")
heatmap(mat,RowSideColors=clust.col[mat.c])
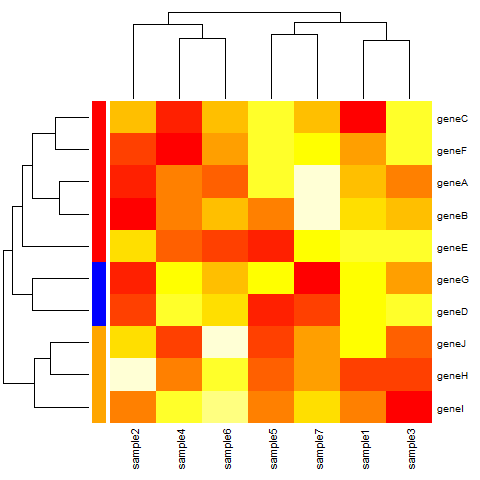
ColSideColorsの使い方も同様です。
なお、heatmap.2のlmatオプションを使用した場合のColSideColors / RowSideColorsの作図順については、以前記事を書きましたのでご参照ください。