IGVのちょっと便利な機能に気づいたのでご紹介します。
ゲノム上の目的の領域の配列を、IGVだけを使って取得してみましょう。
IGVのバージョンは2.3.3で確認しました。
1. BEDで指定したfeatureの配列を取得する
ChIP-seq解析で出力したピーク(BED形式)上の配列を取得したいときの方法です。下図のように、feature上を右クリックし、表示されたメニューから「Copy Sequence」を選択します。
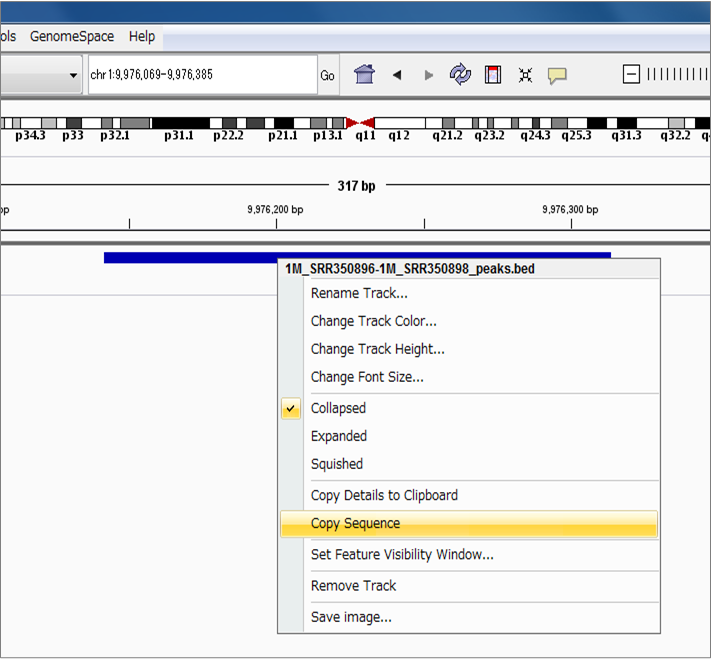
クリップボードに配列情報が保存されますので、使っているワープロソフトやテキストエディタ等にペーストして使用できます。
こちらはわかりやすいので、ご存じの方も多いのではないでしょうか。
2. 任意の領域の配列を取得する
IGVで変異やピークなどのfeatureを眺めながら、「ここからここまでの配列が欲しい!」と思った時。
IGVのツール一覧にあるこちらのアイコンをクリックします。

カーソルが十字になりますので、配列を取り出したい範囲の開始ポジションと終了ポジションをクリックします。これで目的の領域が赤く表示され、選択できたことになります。
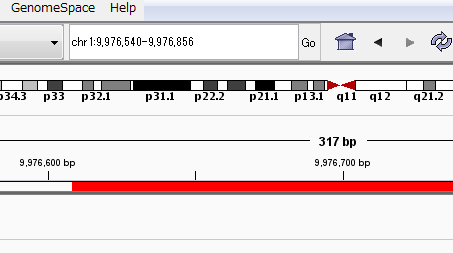
複数個所の配列情報を取り出したい場合は、続けて別の場所を選択できます。
(誤った箇所を選択した場合、赤い部分を右クリックし、メニューから「Delete」を選択することで領域の選択を解除できます)
取り出したい場所を選択し終わったら、ツールバーから「Regions」 > 「Export Regions ...」を選択し、選択した領域を保存します。保存したファイルを、Peakと同じようにIGVで開くことができますので、以降は上記の(1)と同じ方法で配列情報を取り出します。
(参考)
この2番目の方法、最近まで方法を知りませんでした。目視した領域の情報が取得できるので便利です。
(もし、もっと簡単に配列取れる方法あるじゃない! と思った方はぜひ教えてください。情報お待ちしています)