ChIP-seqやRIP-seqでピークが得られた後は、遺伝子をアノテーションしたり、それらの遺伝子がどんなパスウェイに関連するか調べたりしたいですね。
Bioconductorのパッケージseq2pathwayを使って、ゲノム領域に遺伝子をアノテーションしたり、遺伝子にパスウェイをアノテートしたりすることができます。
seq2pathwayのインストールはおなじみbiocLiteで行います。
source("http://bioconductor.org/biocLite.R")
biocLite("seq2pathway")
seq2pathwayパッケージに含まれているテストデータを使ってテストしてみましょう。
library(seq2pathway)
data(Chipseq_Peak_demo)
こんなデータです。
head(Chipseq_Peak_demo)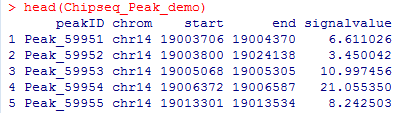
Chipseq_anno <- runseq2gene(inputfile=Chipseq_Peak_demo, genome="hg19", adjacent=F, SNP=F, search_radius=150000, PromoterStop=F, NearestTwoDirection=F)
※実行時にPython2.7が必要というメッセージが出た場合は、https://www.python.org/getit/からお使いの環境に合わせたPython2.7をインストールしてください。
このような結果が得られます。領域に対し、遺伝子のアノテーションがつきました。
head(Chipseq_anno[[1]])