Bioconductorのseq2pathwayパッケージを使ってパスウェイのアノテーションを行います。
※seq2pathwayのインストールはこちらの記事をご覧ください。
seq2pathwayパッケージに含まれているテストデータを使ってテストしてみましょう。
library(seq2pathway)
data(dat_chip)
こんなデータです。遺伝子名とスコアのセットです。
head(dat_chip)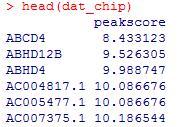
これらの遺伝子に対して、パスウェイをアノテートします。
遺伝子情報は、seq2pathwayに入っているMsigDB_C5データを使います。
data(MsigDB_C5,package="seq2pathway.data")
result_FAIME <- gene2pathway_test="" dat="dat_chip," database="MsigDB_C5," fishertest="T," empiricaltest="T," method="FAIME" alpha="5," logcheck="F," na="" rm="F)</code">
結果は、2つのデータフレームから成るリストです。
names(result_FAIME)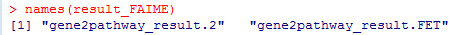
データフレームgene2pathway_result.FETの1列目、3列目、9列目にパスウェイ名、Fisher検定のp値、遺伝子のリストが入っています。
head(result_FAIME$gene2pathway_result.FET[,c(1,3,9)])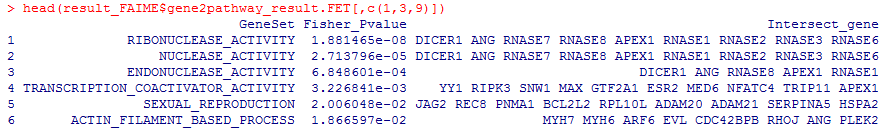
※クリックで拡大します
gene2pathwayではGeneOntologyのアノテーションも行えます。
遺伝子・パスウェイ・GeneOntologyのアノテーションを行うBioconductorのパッケージには、以前紹介したBiomaRtやReactomePAなどさまざまなパッケージがありますが、gene2pathwayも使いやすいと思いました。