
解析ソフトウェア InferCNV は がんおよび正常細胞のシングルセルRNA-seq データから、
コピー数変異を予測することができます。
broad institute が発表しています。
😀 scRNA-seqデータで普通に(?)遺伝子発現比較やクラスタリング解析を解析するのに加えて、同じデータセットでCNVも予測できちゃうなんて、お得ですね!
論文は出ておらず、GitHubのwikiが公式ページのようです。
参考:InferCNV: Inferring copy number alterations from tumor single cell RNA-Seq data Home · broadinstitute/infercnv Wiki · GitHub Bioconductor - infercnv
下に 公式ページのロゴを引用します。
背景のヒートマップは、縦が細胞(さらに太線でサンプル区切り)、 横軸が染色体の位置順に並べられた遺伝子(太線で染色体区切り)
ヒートマップでがん細胞における相対的な遺伝子発現を示しています。
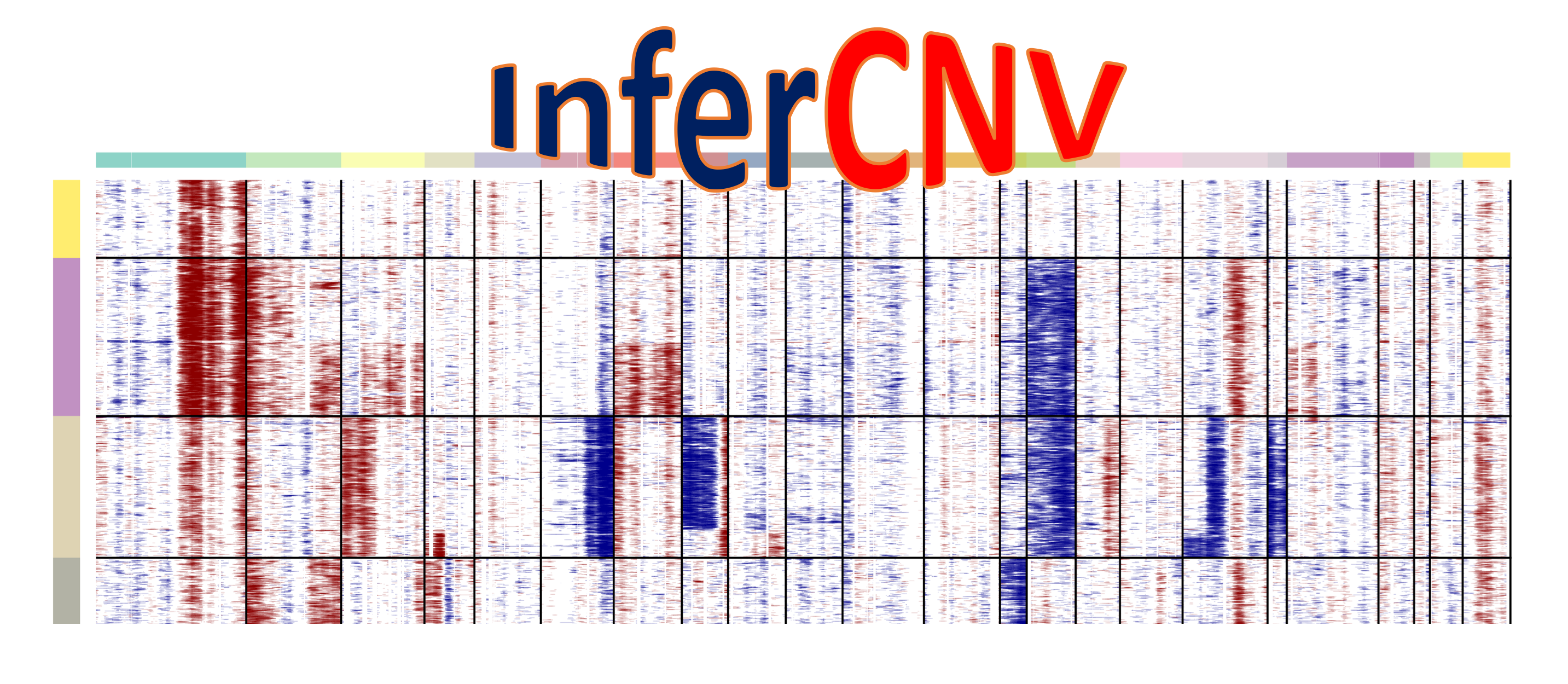
「相対的な遺伝子発現」というのは、
がん細胞の遺伝子発現から 正常細胞の遺伝子発現を引き算して いるということです。
※ 引き算のほかにもセンタリングやノイズ除去が行われます。
その結果、ヒートマップ図を染色体レベルで見て 「chr1の左側、全体的に赤い(= 発現が高い)」⇒ chr1でコピー数増加が起きているのではないか と解釈できます。
もちろん遺伝子を1つづつ見ると、コピー数変異ではなくても がん細胞で高発現/低発現ということはあるでしょうが、
この広い範囲のCNV解析ではあまり気にしなくていい様です。
ヒートマップ可視化に加えて、以下の様にHMMによりコピー数変異の 領域と コピー数を予測できます。
cell_group_name cnv_name state chr start end malignant_MGH36.malignant_MGH36_s1 chr1-region_2 0.5 chr1 3696784 144612683 malignant_MGH36.malignant_MGH36_s1 chr1-region_4 1.5 chr1 151336778 156213123 malignant_MGH36.malignant_MGH36_s1 chr3-region_7 1.5 chr3 3168600 10285427 malignant_MGH36.malignant_MGH36_s1 chr3-region_9 1.5 chr3 45429998 49460186 malignant_MGH36.malignant_MGH36_s1 chr4-region_11 0.5 chr4 53179 187134610 malignant_MGH36.malignant_MGH36_s1 chr5-region_13 1.5 chr5 134181370 177037348 ...
(出力見本:公式サイトより)
必要なデータはこれだけ。簡単ですね。
がん および 正常細胞 の scRNA-seq のrawカウントデータ
それぞれ2検体は必要そうですが、正確な最低個数は確認中。細胞名と「がん/正常」 を対応させるファイル 当然分かることなので、ソフトの指定する形式で書けばよさそうです。
遺伝子の染色体上の位置データ
これは1生物種につき1回用意すれば検体が変わっても同じものを使えそうです。
NGSでCNV検出といえば、ゲノムDNAのデータから分析するイメージでしたが、RNA-seqでも予測はできます。
RNAデータで普通に(?)遺伝子発現比較やクラスタリング解析をするのに加えて、同じデータセットでCNVも予測できちゃうなんて、お得ですね。