Hatena Blogに移行してから初投稿になります。 どうもmisawatです。 お客さん向けのBlogチックなテキストは毎回書いてるんですけどねぇ・・・。(← 言い訳です。はいスイマセン。)
さて、かの有名なメタゲノム解析プログラムQIIMEがメジャーバージョンアップされたので、ちょっと触れたいと思います!
QIIMEとは
QIIMEとはメタゲノム解析を行うためのプログラムが集まったメタゲノム解析ツールです。 主な言語はPythonですが、オープンソースのプログラム、データベース等も多く使われています。 とにかく有名なソフトで論文でも多く見かけます。 かくいう私もQIIMEを使って論文1報出してたりします! ( ̄ー+ ̄)どや

QIIMEについての思い出
このQIIMEですが、正攻法でのインストールがかなーーーり大変です。 QIIMEをダウンロードして、内部的に使われているツールを10個位個別にインストールして、それを全部PATH通して、さらにDBもおとしてアワワワワワ・・・ 少しでも大変さが伝わりましたか? QIIMEそのものというより、QIIMEが動く環境を作るのが大変なんです。 あんまり詳しく書いてないけど、インストール方法のページはこちら
http://qiime.org/install/install.html
バージョンアップ!!
そんなQIIMEがこの度バージョンアップされました!!オオォォォ(゚ロ゚)(゚ロ゚) その名もQIIME2! はい!そのまんまです!

ホームページも新しいものが作られました!
インストール方法
正攻法でインストールすることも出来るのですが、Docker imageがあるので、インストールもとって楽ちんです。 Docker環境が出来ているならば、以下のコマンドを打つだけでインストールできちゃいます。
docker pull qiime2/core:2017.10
ちなみに実行はこんなかんじ。
docker run -t -i -v $(pwd):/data qiime2/core:2017.10 qiime
うん。簡単簡単。
QIIME2へのデータ入力
データのインポートですが、様々な形式に対応しています。以下に羅列してみますと、
- EMP protocol multiplexed single-end fastq
- EMP protocol multiplexed pair-end fastq
- Fastq manifest formats (single-end)
- Fastq manifest formats (pair-end)
- Casava 1.8 single-end demultiplexed fastq
- Casava 1.8 pair-end demultiplexed fastq
- Feature table data
- etc...
今時のシーケンスデータファイルから、旧式のシーケンスデータファイル、QIIME時代の中間フォーマットまで様々な形式でインポートが可能です。 詳しくはこちら!
https://docs.qiime2.org/2017.10/tutorials/importing/
QIIME2のすごい所
QIIME2の凄い所は、GUIのVisualization Toolです!
解析を行っていく中で .qzvファイルというのが出来るのですが、コレを公式のVisualization用ページQIIME 2 View
にアップすると・・・
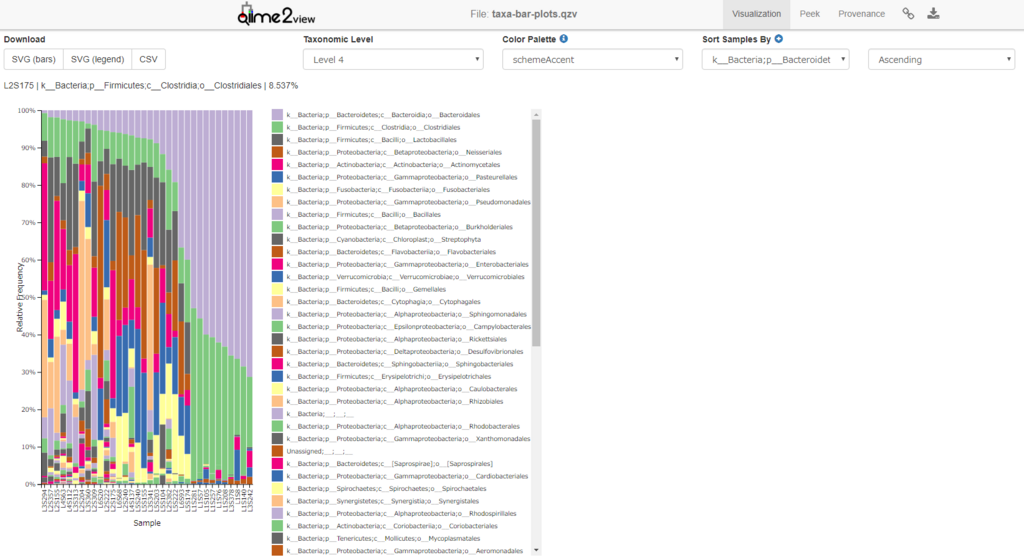
ばばん! これ、GUI操作でグラフの調整が出来るんです。 例えばspecies単位じゃなくて、family単位でみたいなぁとか、そういう変更がやりたい放題。 色もある程度自由にいじれます。
こうやってGUIを全面に押し出すと、我々コマンドライン屋さんは少し抵抗あるかもしれませんが、 CSV形式の出力も出来るので、自分で書いた描画スクリプトと組み合わせるってことも楽々出来ますね!
最後に
とても簡単にでしたがQIIME2について紹介しました! 解析そのものはコマンドライン操作なのですが、かなり簡単に出来るようになったので、 メタゲノム解析を始めたいけど、PC苦手な人でも、結果まで辿り着けそうですね! チュートリアルのページがかなーーり丁寧なので、コレなぞるだけでも良い勉強になりますよ!
https://docs.qiime2.org/2017.10/tutorials/moving-pictures/
好きな言葉は「明日から本気出す」のmisawatがお伝えしました!!
アメリエフでは、バイオデータ解析やそのシステム・インフラ環境の開発に興味のあるエンジニア・リーダー候補を募集しています。
www.wantedly.com