細胞の変化や多様性を調べたい。
そんなあなたにsingle cell RNA-seq解析です。
今回は全長RNAのシーケンシング・解析についてご紹介します。
シングルセル解析のうち、
一般的な、UMIを用いた方法では、RNAの3'または5'末端側に限定してシーケンスすることで、PCRバイアスに影響されずに細胞ごとの遺伝子発現量を調べることができます。
ところが最近、遺伝子発現量だけでなく、RNA, DNA自体に着目した解析が注目されています。
例えば、
- splicing isoform(RNAのスプライシングバリアント/アイソフォーム)
- fusions transcripts (融合遺伝子)
それを可能にするのが全長RNA (実際には逆転写してcDNAになりますが)のシーケンシング・解析です。
シーケンスについて
Smart-seqなどによりシングルセルのライブラリを作成し、シーケンスします。
実際に、シングルセルRNA-Seqでアイソフォームの解析を行い
アイソフォームの発現パターンが異なる細胞群があったという報告も見られます。
シーケンス方式は、ショートリードまたはSingle molecule方式のロングリードに分けられます。
ショートリードの場合、
バーコーディングにより、ショートリードでもアイソフォームの分類が可能です。
(あいまいな部分は分類できない場合もあるようです)
Single moleculeの場合、
シーケンスエラーの問題がありますが、アイソフォームの全長を読むことができます。
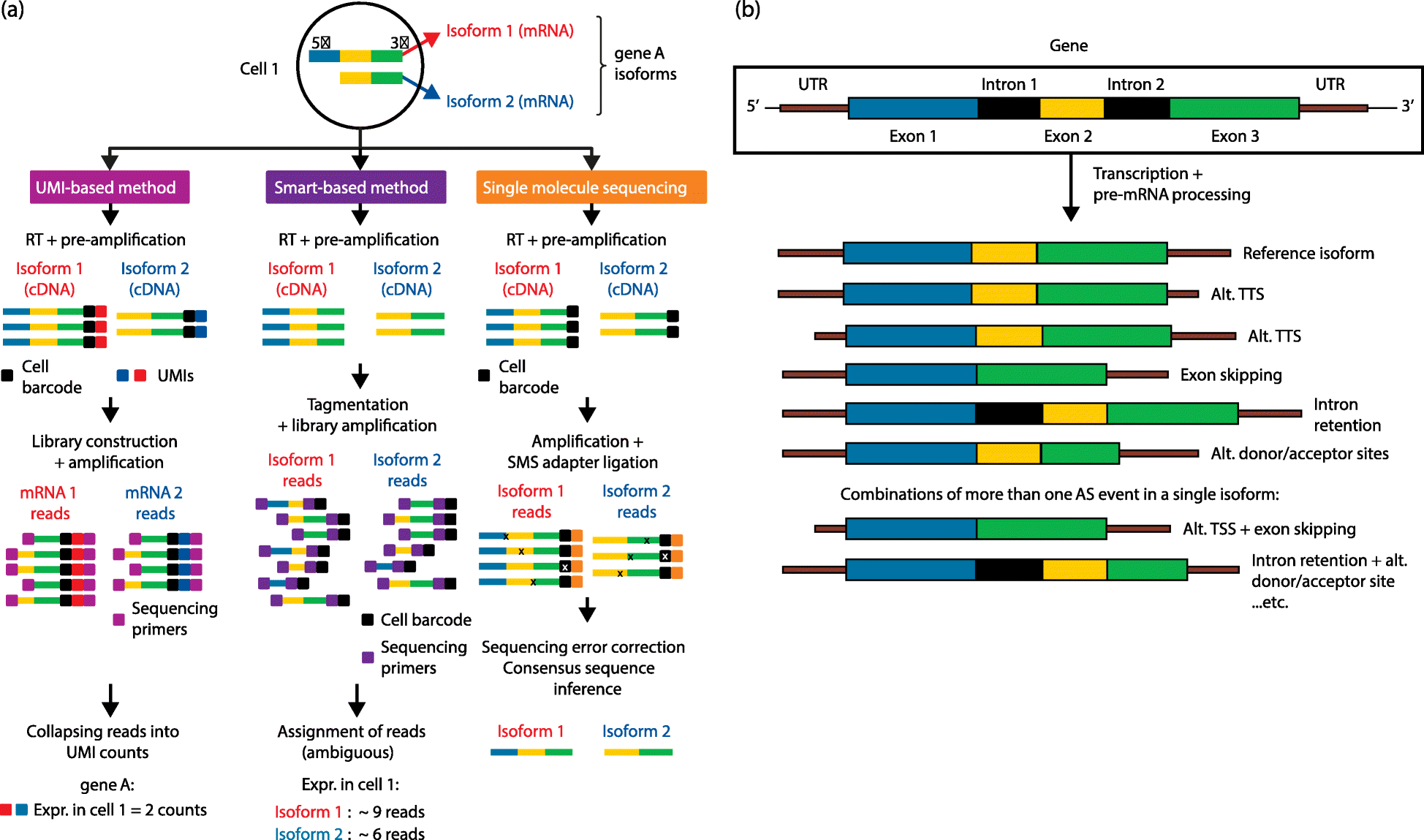 Single-cell RNAseq for the study of isoforms—how is that possible?. Ángeles Arzalluz-Luque et al., Genome Biology 2018. https://doi.org/10.1186/s13059-018-1496-z Fig. 1より
Single-cell RNAseq for the study of isoforms—how is that possible?. Ángeles Arzalluz-Luque et al., Genome Biology 2018. https://doi.org/10.1186/s13059-018-1496-z Fig. 1より
あとがき♪
筆者は違うけど、勝手に以前のブログ↓の続編を書きました。
前回はプロトコル(シングルセルライブラリ作製方法)の分類、
今回はそのうち1つのプロトコル + 様々なシーケンサ を用いたユニークなシーケンス手法でした。
いろいろ書き並べましたが
私は、素直に現代のシーケンス関連技術に感心しました。
細胞1こ1こに分けた上に、RNAを1本も捨てないかのような精密さ。
bulk RNA-seqでできる解析が、どんどんシングルセルRNA-seqでできるようになっているなと思います。