ちょっと前にmiya-yさんがシングルセルATAC-seqについて記事を書いていたので便乗します。 CITE-seq解析について概要と関連ツールのご紹介。
CITE-seqとは
Cellular Indexing of Transcriptomes and Epitopes by Sequencing 略してCITE-seqです。
New York Genome CenterのTechnology Innovation labとSeuratを開発したSatija labとの共同研究により開発されました。
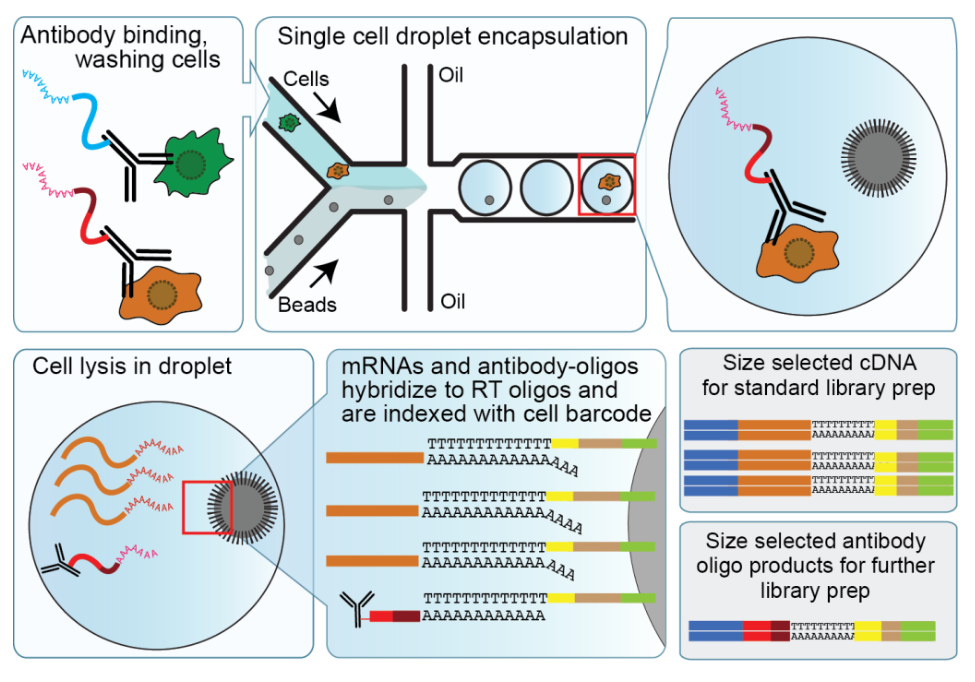
抗体と結合させた、オリゴ(dT)プライマーでラベルした抗体バーコード配列を細胞内RNAと同様に逆転写させることで、RNAの情報と同時に細胞表面のタンパクの情報を統合して解析することができるようなった手法です。
オリゴT(dT)でアニーリングし、ライブラリ調整を行うプロトコル(10x Genomics、 Drop-seq、ddSeqなど)にて、CITE-seqを行うことが可能です。
解析ツール
fastqからカウントマトリックスを作成する際には Christoph Hafemeister氏とPatrick Roelliによって開発されたCITE-seq-Countが利用できます。
また、Satija labのホームページでは、CITE-seqを用いて取得された臍帯血単核細胞 (CBMCs) をSeuratを用いて解析するVignette(Using Seurat with multi-modal data )が公開されています。
解析を行う際はぜひ参考にしましょう!
アメリエフでは、バイオデータ解析やそのシステム・インフラ環境の開発に興味のあるエンジニア・リーダー候補を募集しています。